Congratulations to Rhian Jones and colleagues whose work is in the spotlight of INSB!
Décryptage d’un mécanisme de camouflage du virus du Chikungunya
Un mécanisme complexe de camouflage permet au virus du Chikungunya, un virus pathogène de la famille des alphavirus, de protéger son ARN viral face au système de défense de la cellule. Ce mécanisme est orchestré par la protéine virale nsP1 selon une voie enzymatique spécifique pouvant potentiellement être utiliser pour la mise au point de nouvelles molécules antivirales. Dans cette étude publiée dans la revue PNAS, les scientifiques utilisent une approche par cryomicrocopie électronique pour décrypter ces mécanismes à l’échelle moléculaire et mettre en évidence de nouvelles fonctions pour la protéine nsP1.
La superfamille des alphavirus comprend de nombreux pathogènes dont le virus du Chikungunya. La réplication du génome virale est orchestré par un complexe de quatre protéines virales localisées dans des organelles en forme de panier dérivées de la membrane plasmique de la cellule hôte.
L’une de ces quatre protéines, appelée nsP1, forme une structure poreuse dodécamérique servant de plateforme pour l’assemblage du complexe de réplication. Mais la nsP1 a une fonction enzymatique supplémentaire qui consiste à ajouter une “coiffe” à l’extrémité 5’ des ARN viraux lorsqu’ils sont synthétisés et extrudés de l’organelle. Cette coiffe protège l’ARN viral du système de défense de la cellule hôte.
La mise en place de cette coiffe par la protéine nsP1 se fait selon un mécanisme original et diffèrent de celui utilisé pour les ARN cellulaire, faisant de cette protéine une cible potentiel pour de futurs antiviraux. En particulier, les différentes étapes se font dans un ordre inverse des mécanismes cellulaires. Jusqu’à présent la base moléculaire de ce mécanisme restait insaisissable.
Les scientifiques ont réalisé des analyses biochimiques couplées à des études en cryomicroscopie électronique afin de fournir des informations à haute résolution sur la voie de mise en place de cette coiffe par nsP1.
Le détail de la liaison des substrats dans chacune des 12 poches de la structure dodécamérique expliquent, sur le plan moléculaire, pourquoi les étapes de la mise en place de la coiffe sont inversées par rapport aux enzymes cellulaires. De façon surprenante, les scientifiques ont également démontré que la protéine nsP1 est capable d’éliminer la coiffe de l’ARN. Cette découverte soulève de nombreuses questions sur le rôle de ce nouveau mécanisme mais peut être mis en relation avec l’observation récente d’ARN viraux sans coiffe lors de la libération des virions du Chikungunya.
Les structures obtenues par cryomicroscopie apportent également de nombreuses observations sur la dynamique de la mise en place de la coiffe. Ces détails pourront être exploités pour rechercher de nouveaux inhibiteurs contre l’infection par les alphavirus.
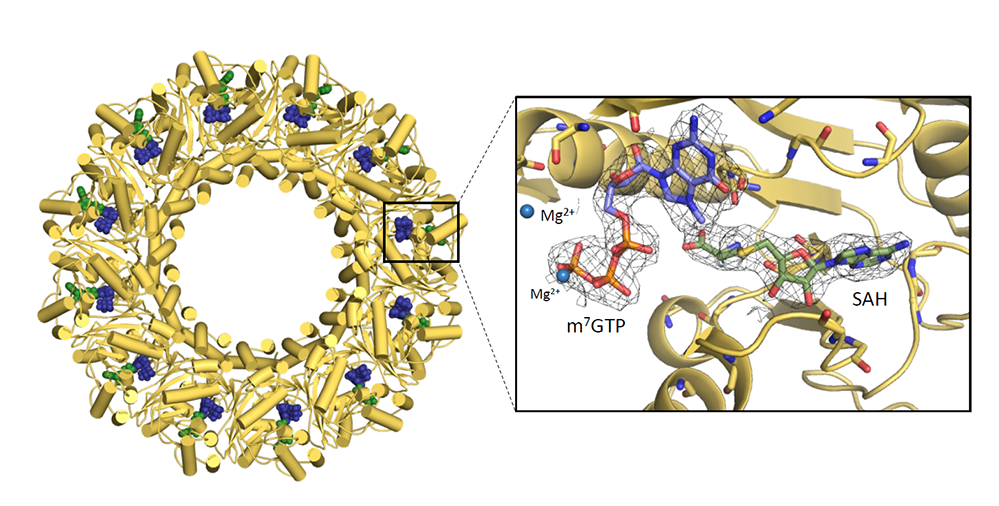
Figure : Structure de la protéine dodécamérique de la protéine nsP1 (jaune) avec les substrats (verts et bleus) sur chacun des 12 sites enzymatiques actifs et détail de l’un des sites.
Pour en savoir plus :
Structural basis and dynamics of Chikungunya alphavirus RNA capping by nsP1 capping pores.
R.Jones, M.Hons, N.Rabah, N.Zamarreño, R.Arranz & J.Reguera.
PNAS, March 13, 2023. DOI: https://doi.org/10.1073/pnas.2213934120
Published on April 18, 2023



