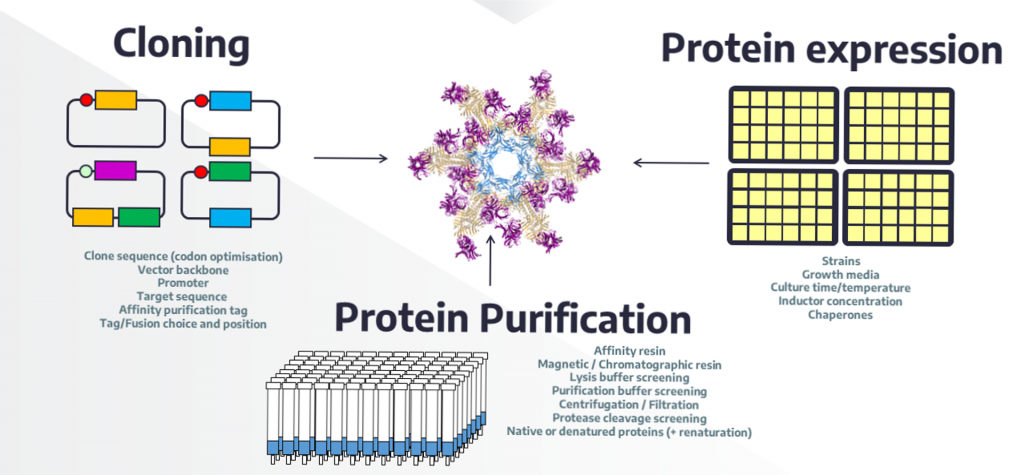
Production dans E. coli, purification et interaction de protéines à haut-débit
Ce service de PBSIM réalise de façon semi-automatique, en utilisant ses propres méthodes à haut-débit, l’ensemble des étapes depuis le clonage des gènes jusqu’aux protéines purifiées.
Son offre évolue et s’enrichit constamment car l’équipe développe des stratégies pour ses propres projets de recherche, ses collaborateurs et ses clients.
Le service de production et purification de protéines est ouvert depuis 2004. Il est reconnu par plusieurs labels nationaux : IBiSA, AMU et FRISBI. Il propose un portfolio de protocoles automatisés (format 96 ou 384) qui couvrent l’ensemble des étapes du clonage, de la production et purification de protéines ainsi que l’étude in vitro des interactions protéine-protéine, protéine-peptide ou protéine-ADN. Depuis son lancement, plus de 10.000 protéines ont été produites pour nos clients, nos collaborateurs ou nos partenaires dans des réseaux nationaux (treize ANR comme partenaire ou sous-contractant) ou internationaux (douze réseaux européens). La plateforme est co-auteure de quatre-vingt publications et enseigne régulièrement certains de ses protocoles dans des formations locales (Polytech, Plinius), nationales (FRISBI, INSERM) et internationales (EMBO).
Contacter le groupe
Services
- Clonage de gènes
- Criblage d’expression : optimisation des conditions de culture pour déterminer et améliorer le niveau de solubilité des protéines dans E. coli
- Renaturation de protéines
- Purification
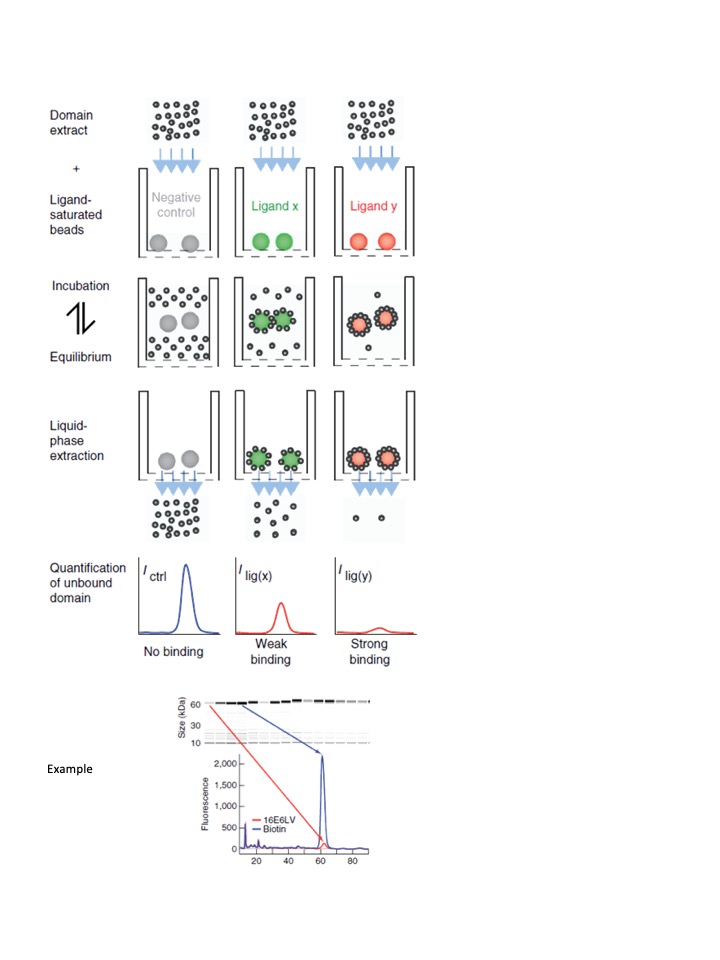
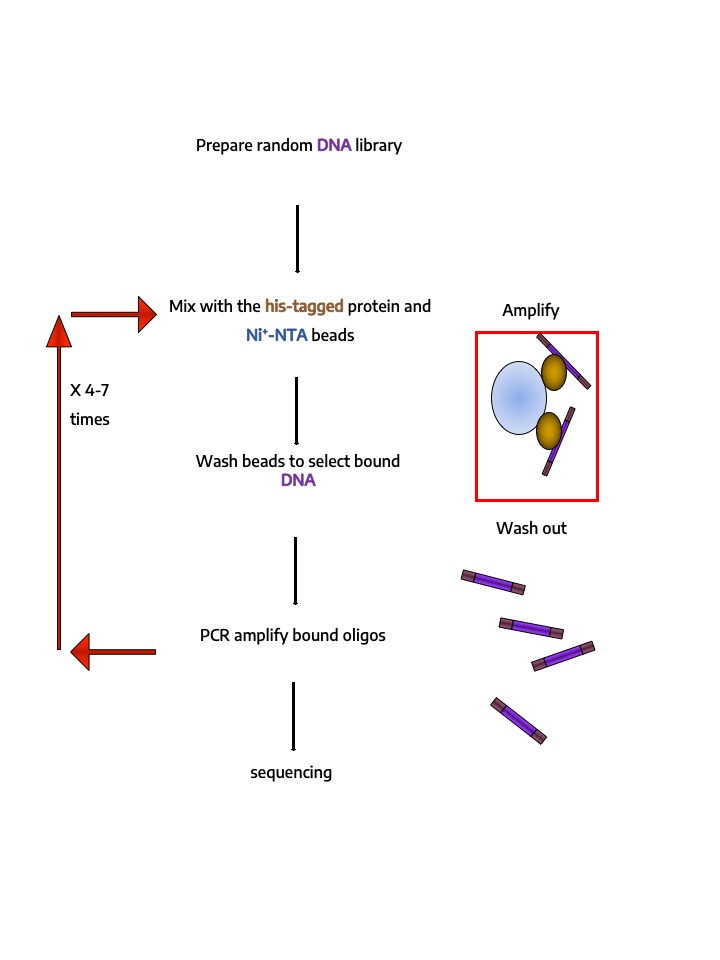
- Identification et quantification in vitro d’interaction protéines-protéines, protéines-peptides (pull-down, holdup, Co-IP) ou protéines-ADN (HT-SELEX) à haut-débit.
Equipement spécifique
- Deux robots de pipetage équipés d’une tête de 96 canaux EVO® (Tecan)
- Une électrophorèses capillaires 96/384 LabChip® GXII Touch (Revvity)
- Un laveur de plaque Hydrospeed® 96/384 avec 4 entrées et module de vide ou magnétique (Tecan)
- Un lecteur de plaque au format 96/384/1536 pour lecture UV/Fluorescence/Luminescence/HTRF Pherastar® FSX (BMG LabTech)
- Deux incubateurs Microtron® pour les cultures bactériennes, capacité de 48 et 72 DeepWells 24/96 (Infors)
- Quatre systèmes de purification (4×4 modules) Aktä®Xpress (Cytiva) à 4°C
- Un automate IDEAL96 pour la purification (ADN et Protéines) sur billes magnétiques (ID solutions)

Développement de méthodes
La très grande majorité des protocoles utilisés pour les prestations de services ont été développées, validées et publiées par la plateforme. Nous développons de nouveaux protocoles quand cela est nécessaire pour nos projets de recherche ou à la demande pour nos clients. Nous collaborons et validons de nouvelles méthodes avec nos collègues du réseau P4EU (>100 plateformes) dont nous sommes un des cinq membres fondateurs.
Tous nos protocoles analytiques sont automatisés en format microplaque 96 ou 384 (robot Tecan) ou sur AKTA Xpress (Cytiva) pour la purification des protéines à plus grande échelle (mg).
Criblage d’expression : optimisation des conditions de culture pour déterminer et améliorer le niveau de solubilité des protéines dans E. coli
Vincentelli et al. (2011) Methods, Vincentelli. R and Romier. C (2013) Curr Opin Struct Biol, N. Saez and Vincentelli. R (2014) Methods Mol Biol.

Renaturation de protéines
Purification de protéines
L’échelle de production et de purification est adaptée selon les besoins du projet:
- Culture et purification « analytique » ou « semi-préparative » : culture en DeepWell 24/96/384 et purification sur plaques filtrantes 24/96/384. Cette échelle est utilisée pour le criblage d’expression ou la production de banques de protéines. La plus grosse banque de protéines purifiée à ce jour comportait 5.000 toxines animales recombinantes.
- Culture et purification « préparative » : culture en fiole de 2 litres et purification sur AKTA Xpress (Cytiva). Toutes les techniques classique de purification sont proposées (affinité, gel-filtration, échange d’ion…). Cette échelle est utilisée pour la purification de projet composé d’une à quelques dizaines de protéines à l’échelle des milligrammes.
Identification et quantification in vitro d’interaction protéines-protéines, protéines-peptides (pull-down, holdup, Co-IP) ou protéines-ADN (HT-SELEX) à haut-débit.
Projets de recherche et collaborations
La plateforme intervient dans des projets de recherche sur fonds privés ou publiques comme partenaire, participant ou prestataire de service. La plupart de ces projets nécessitent des protocoles innovants et c’est le principal moteur de l’évolution des prestations de la plateforme. La plateforme a participé à douze réseaux européens (financements FP5, FP6, FP7, H2020) et treize ANR (l’ANR est l’agence française de financement de la recherche sur projets). La plupart des projets se terminent avec la fin du financement mais certains sont devenus des collaborations sur le long terme financés soit par plusieurs ANR (ou réseaux européens successifs), soit sur fond propre.
Purification de toxines animales
La plateforme a développé depuis 2010 différentes stratégies pour produire des toxines animales sous forme oxydées directement dans E. coli. Ces méthodes ont été raffinées et appliquées à grande échelle sur des milliers de toxines pour le projet VENOMICS (FP7, 2012-2015).
Depuis 2021 la plateforme est un des cinq partenaires européens du projet H2020 Addovenom. Ce projet cherche a développer un nouveau traitement contre les morsures de serpents qui tuent, encore au XXIème siècle, plus de 140.000 personnes par an.
Pour plus d’informations sur la tragédie des morsures de serpents, vous pouvez regardez le documentaire Minutes To Die.
La plateforme est membre du réseau européen des toxines Euven.
Purification et interaction des domaines PDZ
Ce projet découle d’une longue collaboration entre la plateforme et l’équipe du Dr G. Travé à l’IGBMC qui a commencé par l’ANR EPI-HPV-3D (2007-2010) et qui est toujours très active mais financé pour la grande majorité sur fond propre.
Nous avons développé et amélioré ensemble une banque de clones, un protocole de production des domaines PDZ humains ainsi qu’un protocole à haut-débit d’identification et de quantification des interactions des PDZ-PBM pouvant aller jusqu’a la détermination de plusieurs milliers d’affinités/jour et applicable à de très nombreux ensembles protéine-protéine ou protéine-peptide. Cette banque et ces méthodes ont été utilisé par plusieurs équipes européennes notamment par les membres du projet ITN H2020 PDZNet dont la plateforme a été un des laboratoire associé.
Purification de CAZYmes
La plateforme a produit et purifié plus de 2000 CAZYmes pour l’équipe de Glycogénomique (Bernard Henrissat et Nicolas Terrapon) et ses collaborateurs, principalement dans le cadre de trois ANR successives (1000 CAZymes, Pulmarin et ODE) et du projet CAZAI.
Financements en cours
Coordinateur : ANR Full-PDZ-PBM-HPVE6 (2023-2026). Partenaire : Dr. G. Travé at the IGBMC.
Partenaire :
Europe : Addovenom (2021-2025)
NovoNordisk foundation : CAZAI (2023-2027)
INSERM Impact Santé AIR-MT (2025-2027)
ANR AirMN (2021-2025)
ANR PLA2R1 (2026-2028)
Participant : ANR Rha (2024-2027), ODE (2021-2025), DEFINE (2025-2027), ANR PEPLYS 20262029 et AMIDEX GOALS (2024-2028),
Collaboration et prestation
Les collaborations ci dessous correspondent à la période 2016-2021. Quand un lien existe il renvoie à la dernière publication commune sur la période.
Nationale
- Marie-Noëlle Rosso, Giuliano Sciara
- BBF, Marseille
- Rémi Lasserre, Phillipe Naquet, Beatrice NalRogier, Philippe Pierre
- CIML, Marseille
- Jean-Paul Borg, Patrick Fourquet, Pascale Zimmermann
- CRCM, Marseille
- Manos Mavrakis, Jérome Wenger
- Fresnel Institute, Marseille
- André Le Bivic, Laurent Kodjabachian, Alphée Michelot, Aziz Mogrich, Thomas Rival
- IBDM, Marseille
- Félix Rico, Olivier Théodely
- LAI, Marseille
- François Alberto, Tâm Mignot
- LCB, Marseille
- Andreas Zanzoni, Christine Brun
- TAGC, Marseille
- Bruno Coutard
- UVE, Marseille
- Jérome Martin, François Torney
- Biogemma/Limagrain, Chappes
- Nicolas Gilles
- CEA, Saclay
- William Helbert
- Cermav, Grenoble
- Patrick Lemaire
- CRBM, Montpellier
- Stéphanie Cabantous
- CRCT, Toulouse
- Christophe Romier, Arnaud Poterszman, Gilles Travé
- IGBMC, Strasbourg
- Pascale Cossart, Nicolas Wolff
- Pasteur Institute, Paris
- Gérard Lambeau
- IPMC, Valbonne
Internationale
- Kim Remans
- EMBL, Heidelberg, Germany
- Carlos Fontes
- NZYTech, Lisbon, Portugal
- Cherith Reid
- Randox, Crumlin, UK
- Christiane Berger-Schaffitzel, Imre Berger
- University of Bristol, UK
- Henrick Clausen, Kristian Strømgaard
- University of Coppenhagen, Denmark
- Douwe van Sinderen
- University College Cork, Ireland
- László Nyitray
- University Eötvös Loránd, Budapest, Hungary
- Edwin De Pauw, Loic Quinton
- Université de Liège, Belgium
- Stephane Mesnage
- University of Sheffield, UK
- Jonas V. Schaefer
- University of Zurich, Switzerland
- Johnny Habchi
- Wren Therapeutics Ltd, Cambridge, UK
- Robert Allan Heinzen
- NIH Rocky Mountain, Montana, USA
- Sarel-Jacob Fleishman
- Weizmann Institute, Rehovot, Israël
Accessibilité
Académiques et industriels
Coût
A définir en fonction du projet.
Financements








Publications
Le développement de méthodes et le travail effectué sur la plateforme pour nos collaborateurs a donné lieu a plus de quatre-vingt publications comme co-auteur dont des articles dans des journaux à fort facteurs d’impact tels que : Cell, Nature Methods, Nature Catalysis, Nature Microbiology, Nature Communication, P.N.A.S. L’équipe a aussi écrit cinq chapitres de livres et a coordonné l’édition du premier livre de la série « Methods in Molecular Biology » consacré a “High-Throughput Protein Production and Purification « (2019).

Membres
News
September 13, 2022
Year 2 annual meeting in Marseille

October 5, 2021
First ADDovenom annual meeting kicks off in Bristol